- Accueil
- Actualités
- Évolution : Des Mécanismes Révèlent La Proximité Entre Les Eucaryotes et Les Archées
Évolution : des mécanismes révèlent la proximité entre les eucaryotes et les archées
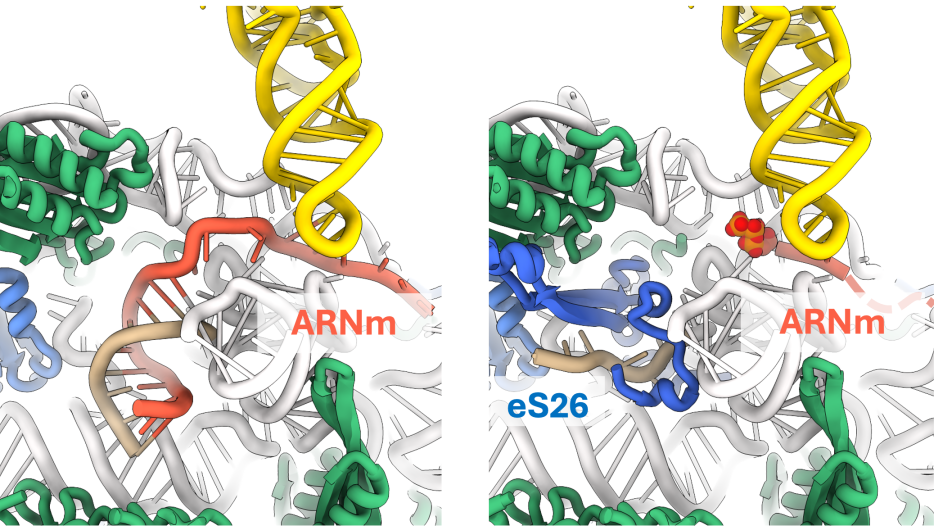 Reconstitution 3D de mécanismes de liaison entre ribosome et ARN messager chez l'archée Saccharolobus solfataricus (Avec et sans séquence "leader", respectivement à gauche et à droite).
Reconstitution 3D de mécanismes de liaison entre ribosome et ARN messager chez l'archée Saccharolobus solfataricus (Avec et sans séquence "leader", respectivement à gauche et à droite).
Parmi les domaines du vivant, entre les bactéries et les eucaryotes (dont les êtres humains font partie) se trouvent les archées. Unicellulaires et sans noyau comme les bactéries, elles sont en revanche beaucoup plus proche des eucaryotes, formés de cellules à noyau, du point de vue de l’évolution. A tel point que les scientifiques ont émis l’hypothèse que c’est à partir d’archées que les eucaryotes seraient apparus. De nombreuses études ont étudié ces liens de parenté en comparant des molécules ou des séquences génétiques présents dans différents organismes. Dans leur publication, l’équipe menée par Emmanuelle Schmitt, directrice de recherche CNRS au BIOC, a emprunté une autre approche, en étudiant les mécanismes de traduction du code génétique en protéines essentielles au fonctionnement de l’organisme.
Au cœur de cette traduction se trouve le ribosome, la machine moléculaire qui, dans la cellule, décode l’information génétique située sur des brins d’ARN messager pour fabriquer les protéines. Les ribosomes sont présents dans toutes les cellules de tous les domaines du vivant, ce qui est idéal pour scruter l’évolution et les liens de parenté. Certaines espèces d’archées sont plus proches des eucaryotes que d’autres. En particulier, le ribosome de l’archée Saccharolobus solfataricus est constitué, entre autres, de trois protéines également présentes chez les eucaryotes.
Observer la liaison entre ARN messager et ribosome
Il existe différentes façons pour que les ARN messagers et les ribosomes se relient. Les ARN messagers qui sont traduits par cette archée se trouvent être de deux types qui se distinguent par l’endroit où commence la séquence à traduire. Dans le premier type d’ARN messager, comme chez les bactéries, cet endroit est précédé par une autre séquence dite « leader ». Mais l’autre type d’ARN messager de cette archée ne possède aucune séquence de ce type. « Comment ces ARN messagers qu’on appelle « leaderless » peuvent-ils être traduit par le ribosome ? Et quelle est la raison de la présence de ces trois protéines supplémentaires ? Ces deux questions nous ont motivé à faire cette étude » explique Emmanuelle Schmitt.
Pour y répondre, les scientifiques ont cultivé en laboratoire Saccharolobus solfataricus, en ont extrait et purifié les ribosomes afin qu’ils restent intacts et actifs. Mélangés à des ARN des deux types, ces « machines à traduire » ont été observés, entre autres, par cryo-microscopie électronique, grâce à la plateforme CIMEX à l’Ecole polytechnique. Les nombreuses images prises permettent de reconstituer en trois dimensions le ribosome en train de se lier aux ARN messagers.
Les scientifiques ont ainsi vu la coexistence de deux mécanismes de liaison. Les ARN messagers avec une séquence « leader » peuvent lier cette séquence avec une extrémité du ribosome de façon robuste, comme dans le cas des bactéries. En revanche, pour lier les ARN de type « leaderless », cette extrémité peut être « bloquée » par une protéine (appelée eS26) que ces archées ont en commun avec les eucaryotes. « Nous pensons que c’est la concentration de cette protéine qui permet de passer d'un mode de liaison à un autre et donc de réguler la traduction » précise la chercheuse. Ce second mécanisme de liaison des ARN messagers pourrait être une version très préliminaire de celui observé chez les eucaryotes. La présence de ces deux processus, l’un proche de celui des bactéries, l’autre proche de celui des eucaryotes, conforte l'hypothèse que ces derniers auraient pu évoluer à partir d’archées.

En présence d'un ARN messager avec séquence leader (en rouge, à gauche), cette dernière se lie à une extrémité du ribosome de l'archée (en marron). La protéine eS26 (en bleu, à droite) peut s'enrouler autour de cette extrémité du ribosome pour la neutraliser, permettant la liaison avec des ARN messagers "leaderless".
La publication scientifique :
Bourgeois, G., Coureux, PD., Lazennec-Schurdevin, C. et al. Structures of Saccharolobus solfataricus initiation complexes with leaderless mRNAs highlight archaeal features and eukaryotic proximity. Nat Commun 16, 348 (2025). https://doi.org/10.1038/s41467-024-55718-5
*BIOC : une unité mixte de recherche CNRS, École polytechnique, Institut Polytechnique de Paris, 91120 Palaiseau, France
*LCM : une unité mixte de recherche CNRS, École polytechnique, Institut Polytechnique de Paris, 91120 Palaiseau, France
 Je soutiens l'X
Je soutiens l'X